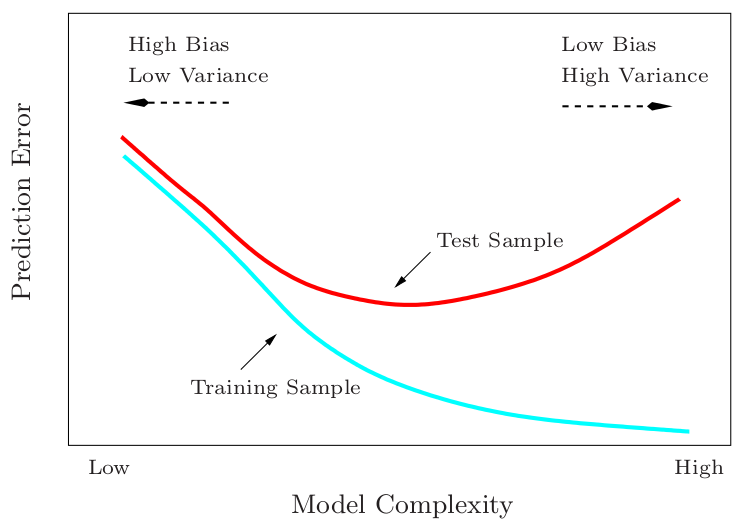
03.01 - MODELS FROM DATA#
!wget --no-cache -O init.py -q https://raw.githubusercontent.com/rramosp/ai4eng.v1/main/content/init.py
import init; init.init(force_download=False); init.get_weblink()
'20201.ai4eng'
1. Un modelo para datos (distribuciones) 1D#
Caso ideal: Sabemos las distribuciones de las que vienen los datos. \(\Rightarrow\) podemos calcular analíticamente nuestro modelo.
¿Podemos encontrar un modelo con 100% de acierto? ¿Por qué sí, o por qué no?
from local.lib import mlutils
from scipy import stats
from scipy import optimize
import numpy as np
import matplotlib.pyplot as plt
from sklearn.metrics import accuracy_score
from sklearn.model_selection import train_test_split
from sklearn.linear_model import LogisticRegression
from sklearn.tree import DecisionTreeClassifier
from sklearn.ensemble import RandomForestClassifier
from sklearn.svm import SVC
import warnings
warnings.filterwarnings("ignore")
%matplotlib inline
## KEEPOUTPUT
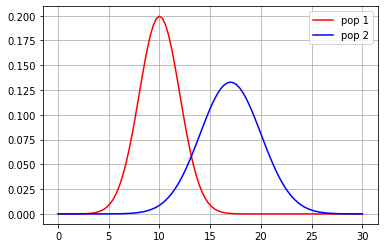
d1 = stats.norm(loc=10,scale=2)
d2 = stats.norm(loc=17,scale=3)
x = np.linspace(0,30,100)
plt.plot(x, d1.pdf(x), color="red", label="pop 1")
plt.plot(x, d2.pdf(x), color="blue", label="pop 2")
plt.grid()
plt.legend();
## KEEPOUTPUT
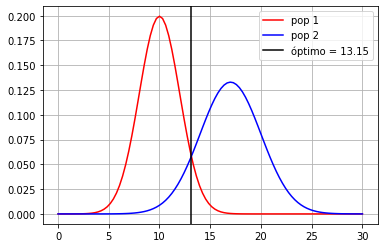
x = np.linspace(5,25,1000)
minx = x[np.argmin(np.abs(d1.pdf(x)-d2.pdf(x)))]
print("frontera óptima en %.2f"%minx)
x = np.linspace(0,30,100)
plt.plot(x, d1.pdf(x), color="red", label="pop 1")
plt.plot(x, d2.pdf(x), color="blue", label="pop 2")
plt.axvline(minx, color="black", label="óptimo = %.2f"%minx)
plt.grid()
plt.legend();
frontera óptima en 13.15
cálculo analítico de los errores de clasificación#
## KEEPOUTPUT
print("pop 1 error", 1-d1.cdf(minx))
print("pop 2 error", d2.cdf(minx))
pop 1 error 0.05773516274534907
pop 2 error 0.09957961739117976
Caso real: Tenemos una muestra de los datos
\(\rightarrow\) ¿Cómo determinamos donde poner la frontera?
\(\rightarrow\) ¿Qué frontera qusiéramos obtener?
## KEEPOUTPUT
from local.lib import mlutils
mlutils.show_1D_dataset_samples(100, d1, d2, n_datasets=1, dot_alpha=.5, line_alpha=0, figsize=(20,1))
plt.axis("on"); plt.ylim(.095, .105); plt.yticks([]);
plt.axhline(.1, color="black", alpha=.2)
<matplotlib.lines.Line2D at 0x7f91456ee690>

## KEEPOUTPUT
mlutils.show_1D_dataset_samples(10, d1, d2, dot_alpha=.3)
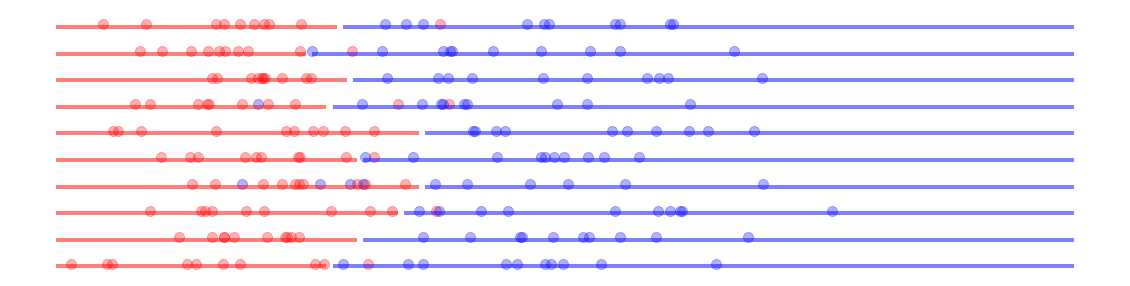
## KEEPOUTPUT
mlutils.show_1D_dataset_samples(100, d1, d2, dot_alpha=.05)

## KEEPOUTPUT
mlutils.show_1D_dataset_samples(10000, d1, d2, dot_alpha=.001)

2. Un modelo para datos (distribuciones) 2D#
en 2D, un modelo de clasificación es una frontera en el plano
supongamos que tenemos acceso a las distribuciones de las que surgen los datos \(\rightarrow\) podemos muestrear tantas veces como queramos
¿cuál es la frontera que produce menos error de clasificación?
\(\epsilon\) es el error de clasificación calculado analíticamente con la frontera óptima porque conocemos las distribuciones que generan los datos.
\(\hat{\epsilon}\) es el error de clasificacón calculado con la muestra de datos y con la frontera óptima (conocida también como frontera bayesiana).
## KEEPOUTPUT
mc = mlutils.Example_Bayes2DClassifier(mean0=[1.5, 2.5], cov0=[[0.1, 0.], [0., 0.1]],
mean1=[1.5, 2.], cov1=[[0.2,0.1],[0,.2]])
X,y = mc.sample(500)
mlutils.plot_2Ddata_with_boundary(mc.predict, X, y, line_width=3, line_color="green", dots_alpha=.3)
plt.title(" $\hat{\epsilon}=%.3f$"%(1-mc.score(X,y))+" $\epsilon=%.3f$"%(1-mc.analytic_score()));
plt.grid();
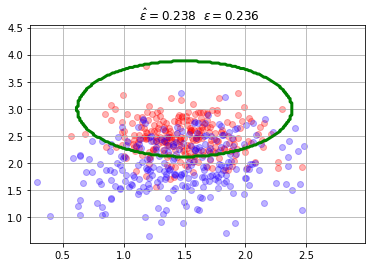
haz los siguientes experimentos:
separa los centros de las distribuciones de cada clase (
mean0,mean1).aumenta y disminuye las matrices de covarianza.
aumenta y disminuye el número de muestras.
observa la estabilidad de \(\hat{\epsilon}\) respecto a \(\epsilon\) según ambas clases están más mezcladas o hay más o menos datos.
en general SOLO TENEMOS UNA MUESTRA de los datos, porque no tenemos conocimiento de las distribuciones que los generan. Los métodos de estadística y de machine learning están diseñados para esta situación.
Ejecuta la siguiente celda y pregúntate cada vez, ¿qué frontera establecerías con los datos que ves?.
Fíjate que tenemos distintas muestras de una misma disitribución de base. Es decir, la realidad detrás de estos datos siempre es la misma.
Aumenta el número de muestras y hazte cada vez la misma pregunta.
## KEEPOUTPUT
X,y = mc.sample(300)
mlutils.plot_2Ddata(X, y, dots_alpha=.3)
plt.grid()
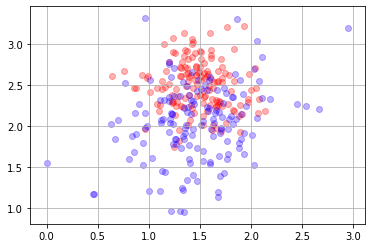
Los algoritmos de machine learning:#
Los algoritmos de clasificación calculan fronteras entre los datos.
Parten de una muestra de los datos, no de las distribuciones.
Por tanto, no conocemos la forma de la frontera bayesiana.
O sea, partimos ciegos!!!!!!!
Para abordar esta situación, cualquier algoritmo tiene necesariamente que plantear una alguna suposición de base:
los datos vienen de distribuciones normales.
las columnas son estadísticamente independientes.
la frontera es lineal, o cuadrática.
la frontera se representa con una red neuronal.
Teniendo esto en cuenta, y partiendo de una muestra de los datos:
el objetivo de un usuario de ML es acercarse lo más posible a la frontera bayesiana (que no sabemos cómo es).
distintos algoritmos ML tienen distintas capacidades para modelar fronteras (un clasificador lineal no puede captuarar una frontera cuadrática).
necesitamos dos tipos de herramientas:
una buena colección de algoritmos ML.
métodos para saber qué tan cerca estamos de la frontera bayesiana.
observa cómo un clasificador lineal aproxima la frontera con diferentes tamaños de muestras#
cambia el parámetro
n_samplesy experimenta con el siguiente código.usa luego estimadores distintos. P.ej.
estimator = SVC(gamma=1) estimator = RandomForestClassifier() estimator = SVC(gamma=100)
Hazte las siguientes preguntas:
¿qué complejidad es capaz de capturar un estimador?
## KEEPOUTPUT
n_samples = 50
estimator = LogisticRegression()
estimator = SVC(gamma=100)
plt.figure(figsize=(15,3))
for i in range(3):
plt.subplot(1,3,i+1)
mlutils.plot_estimator_border(mc, estimator, mins=[0,-1], maxs=[3,4], n_samples=n_samples, legend=False)
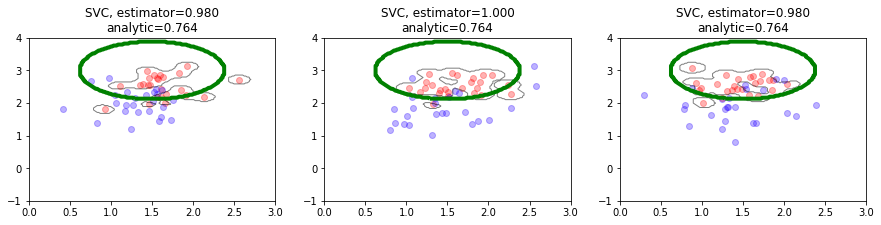
hacemos ahora el experimento más exhaustivo repitiendo 20 veces el siguiente proceso:
se muestrea el dataset (recuerda que excepcionalmente tenemos acceso a las distribuciones que generan los datos y por tanto podemos hacer esto).
se calcula la fronter de clasificación obtenida por el estimador usando el dataset muestreado.
se pinta esta frontera (en negro).
en verde se muestra la frontera bayesiana.
## KEEPOUTPUT
mlutils.sample_borders(mc, estimator, samples = [10,50,100,500], n_reps=20, mins=[0,-1], maxs=[3,4])
100% (4 of 4) |##########################| Elapsed Time: 0:00:09 Time: 0:00:09

3. Solo tenemos un dataset#
remuestrea, reentrena para medir el desempeño y entender la estabilidad
prueba con
test_pctentre 0.1 y 0.9. observa la divergencia entre train y test cuandotest_pctes más cercano a 0.9. ¿por qué?prueba con
SVC(gamma=100). En este caso observa la divergencia entre train y test incluso cuandotest_pctes pequeño. ¿por qué?prubea además con
dataset_sizeentre 20 y 500
Entiende la diferencia entre tener pocos datos y overfitting
Configuraciones interesantes sobre
mc = mlutils.Example_Bayes2DClassifier(mean0=[1.5, 1.5], cov0=[[0.5, 0.1], [0.3, 0.5]],
mean1=[1.2, 2.], cov1=[[0.2,0.1],[0,.5]])
SVC \(\gamma=100\),
dataset_size=500(overfitting con pocos datos, convergencia con muchos)SVC \(\gamma=100\),
dataset_size=2000(overfitting con pocos datos, convergencia con muchos)SVC \(\gamma=.01\),
dataset_size=100(variabilidad siempre, convergencia promedio con pocos datos)SVC \(\gamma=1\),
dataset_size=100(variabilidad siempre, convergencia promedio con pocos datos)LogisticRegression,
dataset_size=100(nunca converge a la frontera bayesiana)
## KEEPOUTPUT
dataset_size = 200
mc = mlutils.Example_Bayes2DClassifier(mean0=[1.5, 1.5], cov0=[[4., 0.5], [0.1, 4.]],
mean1=[1.5,4.], cov1=[[1,0.5],[0.1,1.]])
X,y = mc.sample(dataset_size)
mlutils.plot_estimator_border(mc, n_samples=dataset_size, legend=False)
analitic_score = mc.analytic_score()
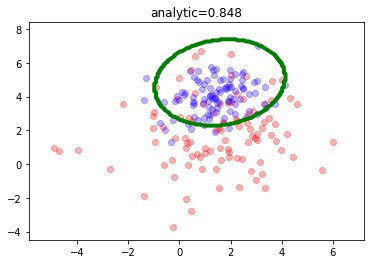
realizamos un muestreo para dividir entre train y test
## KEEPOUTPUT
test_pct = .3
X_train, X_test, y_train, y_test = train_test_split(X,y,test_size=test_pct)
print(X_train.shape, y_train.shape)
print(X_test.shape, y_test.shape)
(140, 2) (140,)
(60, 2) (60,)
## KEEPOUTPUT
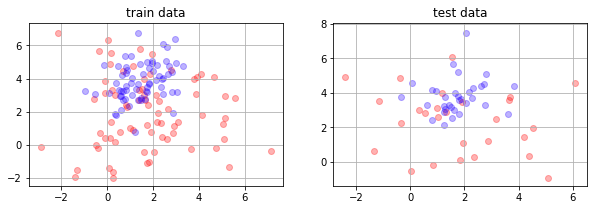
plt.figure(figsize=(10,3))
plt.subplot(121)
mlutils.plot_2Ddata(X_train, y_train, dots_alpha=.3)
plt.title("train data")
plt.grid()
plt.subplot(122)
mlutils.plot_2Ddata(X_test, y_test, dots_alpha=.3)
plt.grid()
plt.title("test data")
Text(0.5, 1.0, 'test data')
#estimator = SVC(gamma=1)
estimator = SVC(gamma=100)
#estimator = LogisticRegression()
#estimator = RandomForestClassifier()
estimator.fit(X_train, y_train)
SVC(C=1.0, cache_size=200, class_weight=None, coef0=0.0,
decision_function_shape='ovr', degree=3, gamma=100, kernel='rbf',
max_iter=-1, probability=False, random_state=None, shrinking=True,
tol=0.001, verbose=False)
## KEEPOUTPUT
print("accuracy train %.2f"%estimator.score(X_train,y_train))
tr_preds = estimator.predict(X_train)
print("predicciones para train")
print(tr_preds)
print("ground truth para train")
print(y_train)
accuracy train 1.00
predicciones para train
[1. 0. 0. 1. 0. 1. 1. 0. 0. 0. 0. 1. 1. 0. 0. 1. 0. 0. 0. 0. 0. 0. 0. 1.
0. 1. 1. 0. 0. 0. 1. 0. 1. 0. 1. 0. 0. 1. 1. 1. 0. 0. 0. 0. 0. 1. 1. 1.
1. 1. 0. 0. 0. 1. 1. 1. 1. 0. 1. 0. 1. 0. 1. 0. 1. 0. 0. 0. 1. 0. 0. 1.
1. 1. 1. 0. 1. 1. 1. 0. 0. 0. 0. 1. 1. 0. 0. 0. 1. 0. 0. 1. 1. 1. 0. 0.
1. 1. 0. 0. 1. 1. 1. 0. 0. 1. 0. 0. 0. 1. 1. 0. 1. 1. 1. 0. 1. 1. 0. 1.
0. 0. 1. 0. 1. 1. 0. 1. 1. 1. 0. 0. 0. 1. 1. 1. 1. 1. 0. 0.]
ground truth para train
[1. 0. 0. 1. 0. 1. 1. 0. 0. 0. 0. 1. 1. 0. 0. 1. 0. 0. 0. 0. 0. 0. 0. 1.
0. 1. 1. 0. 0. 0. 1. 0. 1. 0. 1. 0. 0. 1. 1. 1. 0. 0. 0. 0. 0. 1. 1. 1.
1. 1. 0. 0. 0. 1. 1. 1. 1. 0. 1. 0. 1. 0. 1. 0. 1. 0. 0. 0. 1. 0. 0. 1.
1. 1. 1. 0. 1. 1. 1. 0. 0. 0. 0. 1. 1. 0. 0. 0. 1. 0. 0. 1. 1. 1. 0. 0.
1. 1. 0. 0. 1. 1. 1. 0. 0. 1. 0. 0. 0. 1. 1. 0. 1. 1. 1. 0. 1. 1. 0. 1.
0. 0. 1. 0. 1. 1. 0. 1. 1. 1. 0. 0. 0. 1. 1. 1. 1. 1. 0. 0.]
## KEEPOUTPUT
print("\naccuracy test %.2f"%estimator.score(X_test,y_test))
ts_preds = estimator.predict(X_test)
print("predicciones para test")
print(ts_preds)
print("ground truth para test")
print(y_test)
accuracy test 0.65
predicciones para test
[1. 0. 0. 0. 1. 1. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 1.
1. 0. 0. 0. 1. 1. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 1. 1. 0. 0. 0.
1. 0. 0. 0. 1. 0. 0. 1. 0. 0. 0. 1.]
ground truth para test
[1. 0. 1. 1. 1. 1. 0. 0. 0. 0. 0. 1. 1. 0. 0. 0. 1. 0. 0. 1. 1. 1. 1. 1.
1. 0. 0. 0. 1. 1. 0. 0. 1. 0. 1. 0. 0. 1. 1. 1. 0. 0. 0. 1. 1. 1. 0. 1.
1. 0. 1. 0. 0. 0. 1. 1. 1. 1. 0. 1.]
## KEEPOUTPUT
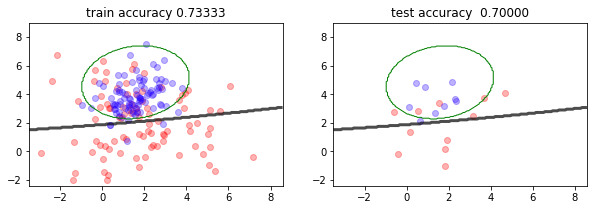
estimator = SVC(gamma=.001)
X_train, X_test, y_train, y_test = train_test_split(X,y,test_size=.1)
estimator.fit(X_train, y_train)
trsc = estimator.score(X_train, y_train)
tssc = estimator.score(X_test, y_test)
print("train_score %5.2f"%estimator.score(X_train, y_train))
print("test_score %5.2f"%estimator.score(X_test, y_test))
plt.figure(figsize=(10,3))
plt.subplot(121)
mlutils.plot_2Ddata(X_train, y_train, dots_alpha=.3)
mlutils.plot_2D_boundary(estimator.predict, np.min(X, axis=0), np.max(X, axis=0),
line_width=3, line_alpha=.7, label=None)
plt.title("train accuracy %.5f"%estimator.score(X_train, y_train))
mlutils.plot_2D_boundary(mc.predict, np.min(X, axis=0), np.max(X, axis=0),
line_width=1, line_alpha=1., line_color="green", label="bayes boundary")
plt.subplot(122)
mlutils.plot_2Ddata(X_test, y_test, dots_alpha=.3)
mlutils.plot_2D_boundary(estimator.predict, np.min(X, axis=0), np.max(X, axis=0),
line_width=3, line_alpha=.7, label=None)
plt.title("test accuracy %.5f"%estimator.score(X_test, y_test))
mlutils.plot_2D_boundary(mc.predict, np.min(X, axis=0), np.max(X, axis=0),
line_width=1, line_alpha=1., line_color="green", label="bayes boundary")
train_score 0.73
test_score 0.70
(0.85395, 0.14605)
4. Learning curves#
normalmente tenemos datasets de muchas dimensiones (columnas) y no podemos visualizar los datos como en 2D \(\rightarrow\) necesitamos métodos para recabar evidencia sobre si tenemos pocos datos, estamos haciendo overfitting, etc.
Las curvas de aprendizaje nos ayudan a esto.
Realiza el experimento desde la sección anterior, con distintos tamaños del dataset inicial y con distintos algoritmos.
X,y = mc.sample(500)
## KEEPOUTPUT
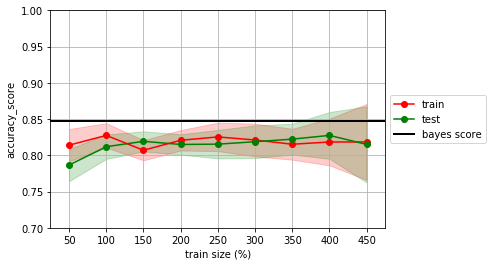
estimator = LogisticRegression()
mlutils.lcurve(estimator, X, y, n_reps=20, score_func=accuracy_score)
plt.axhline(analitic_score, lw=2, color="black", label="bayes score")
plt.legend(loc='center left', bbox_to_anchor=(1, 0.5))
plt.ylim(0.7,1.0);
100% (9 of 9) |##########################| Elapsed Time: 0:00:00 Time: 0:00:00
## KEEPOUTPUT
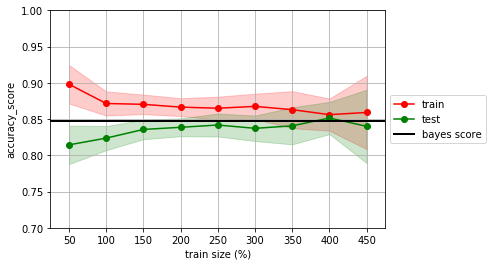
estimator = SVC(gamma=1)
mlutils.lcurve(estimator, X, y, n_reps=20, score_func=accuracy_score)
plt.axhline(analitic_score, lw=2, color="black", label="bayes score")
plt.legend(loc='center left', bbox_to_anchor=(1, 0.5))
plt.ylim(0.7,1.0);
100% (9 of 9) |##########################| Elapsed Time: 0:00:00 Time: 0:00:00
## KEEPOUTPUT
estimator = SVC(gamma=100)
mlutils.lcurve(estimator, X, y, n_reps=20, score_func=accuracy_score)
plt.axhline(analitic_score, lw=2, color="black", label="bayes score")
plt.legend(loc='center left', bbox_to_anchor=(1, 0.5))
plt.ylim(0.4,1.0);
100% (9 of 9) |##########################| Elapsed Time: 0:00:00 Time: 0:00:00
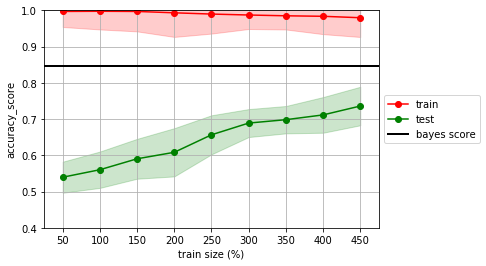
no olvides que normalmente no conocemos la frontera bayesiana, y por tanto no tenemos el bayes score
Taxonomía de problemas de machine learning#
Supervisados
Clasificación
Regresión
No supervisados
Agrupamiento
Estimación de densidad
etc.
Reinforcement learning